文章信息
文章题目:Amplification editing enables efficient and precise duplication of DNA from short sequence to megabase and chromosomal scale
期刊:Cell
发表时间:2024年6月26日
主要内容:武汉大学医学研究院、中南医院医学研究院、教育部免疫与代谢前沿科学中心、泰康生命医学中心殷昊教授课题组在Cell期刊发表题为Amplification editing enables efficient and precise duplication of DNA from short sequence to megabase and chromosomal scale的研究性论文。本项工作研发了一种名为Amplification Editing(AE)的方法,以可编程的方式精确高效地复制从小片段到包含特定染色体大部分区域的基因组序列,是一种高效精确的基因组结构编辑工具,将精准复制的范围从单个基因位点扩展到染色体层面。
原文链接:https://doi.org/10.1016/j.cell.2024.05.056
使用TransGen产品:
pEASY®-Basic Seamless Cloning and Assembly Kit (CU201)
Trans1-T1 Phage Resistant Chemically Competent Cell (CD501)

研究背景
基因扩增(基因重复)是指在生物体内复制DNA片段,形成重复序列。扩增范围从几个碱基对到染色体大部分区域。基因扩增是一种染色体的结构变异,在进化、遗传性疾病和癌症的发生发展中扮演重要角色。现有的基因编辑工具能做到单个位点的精准编辑,但是缺乏高效精确的基因组结构编辑工具。
文章概述
首先,课题组在11个细胞系的多个位点进行了验证。在AE复制长度和效率方面,当复制的DNA长度为20 bp至8 Kb时,AE可进行多次复制,形成串联重复序列。当复制长度为1 Mb时,AE的编辑效率最高达73.0%;复制长度为100 Mb(接近人类染色体的平均长度)时,效率达到3.4%。通过原位荧光杂交、核型分析、全基因组测序等实验,可以直接观察到染色体区域的复制。在AE编辑的精准度方面,连接处的二代测序和全长的三代测序数据显示,indels均低于1%。
其次,课题组利用AE精准修正了绿色荧光蛋白序列和实现miRNA的内源性过表达。同时,课题组在髓性白血病细胞系K562中建立了α地中海贫血症模型,并测试了AE的效果,实现了HBA基因的mRNA水平的提高和 α/γ-珠蛋白比例的上升。这些结果表明AE能够恢复基因表达、扩增miRNA并增加模型细胞系中的α-珠蛋白的表达。
接下来,AE在人源和鼠源干细胞的多个位点均实现了高效精准的短片段和Mb级别编辑,模拟了染色体微重复疾病的基因型,并使用全基因组测序验证了编辑的精准性。这表明AE能够精准构建超大片段复制相关的疾病模型。
最后,该研究对AE的机制进行了初步探究。在被抑制细胞周期的RPE-1细胞中,AE的效率降低。在小鼠原代神经元中,AE只可实现短片段的复制。这说明AE在Mb级别的复制可能依赖于细胞周期。
综上所述,该研究开发了一种名为“Amplification Editing(AE)”的基因组编辑工具,可实现从短片段到染色体长度的精准复制。该工具的开发可促进基因组疾病模型的建立,推动基因进化和癌症机制相关的研究。
全式金生物产品支撑
优质的试剂是科学研究的利器。全式金生物无缝克隆试剂盒pEASY®-Basic Seamless Cloning and Assembly Kit (CU201)和Trans1-T1 克隆感受态细胞Trans1-T1 Phage Resistant Chemically Competent Cell (CD501)助力本研究。
pEASY®-Basic Seamless Cloning and Assembly Kit (CU201)
本产品利用特殊的重组酶和同源重组的原理,可以将任意方法线性化后的载体和与其两端具有15-25 bp重叠区域的PCR片段定向重组,可以实现1-5个片段的高效无缝拼接。
产品特点:
● 快速:仅需要5~15分钟反应时间。
● 简单:不受片段酶切位点的影响,无需对片段酶切。
● 高效:阳性率达95% 以上。
● 无缝:不引入额外的序列。
实验数据
单片段连接
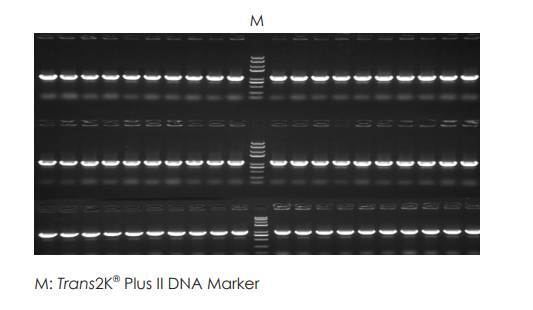
使用 TransGen 产品,将 5 个不同长度片段(0.5 kb,1.2 kb,1.4 kb,1.8 kb,3.9 kb)混合后插入载体中,酶切鉴定插入效果。结果表明,不同长度的片段均正确插入载体,成功进行多片段连接。
多片段连接
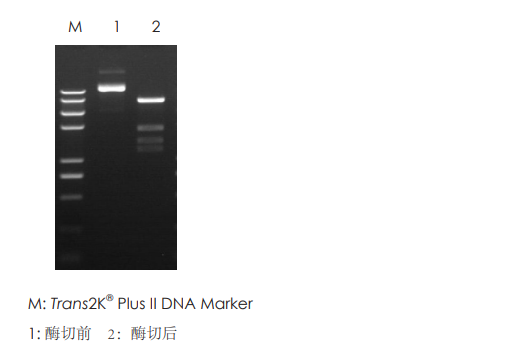
1 kb PCR片段插入pUC19载体后,PCR鉴定插入效果。结果显示,插入片段的阳性率达 100%。
Trans1-T1 Phage Resistant Chemically Competent Cell (CD501)
本产品经特殊工艺制作,可用于DNA的化学转化。使用pUC19质粒DNA检测,转化效率高达109 cfu/μg DNA以上。
产品特点:
● Trans1-T1 Phage Resistant感受态细胞是目前生长速度最快的感受态细胞,在氨苄青霉素平板上,8-9 小时可见克隆。
● 用于蓝、白斑筛选,12 小时可见蓝斑。
● 将过夜培养的单克隆在2 mL的LB培养基中培养4-5小时即可进行小量质粒提取。
● 适用于高效的DNA 克隆和质粒扩增,减少克隆DNA同源重组的发生,提高质粒DNA的产量和质量。
● 具有T1,T5噬菌体抗性。
全式金生物产品再一次登上Cell期刊,证明了大家对全式金生物产品品质和实力的认可,也完美诠释了全式金生物一直以来秉承的“品质高于一切,精品服务客户”的理念。全式金生物始终在助力科研的道路上砥砺前行,希望未来能与更多的科研工作者并肩奋斗,用更多更好的产品持续助力科研。
使用pEASY®-Basic Seamless Cloning and Assembly Kit (CU201) 产品发表的部分文章:
• Zhang R W, Zhou H, et al. Amplification editing enables efficient and precise duplication of DNA from short sequence to megabase and chromosomal scale [J]. Cell, 2024.
• Zhang Y , Dai F , Chen N ,et al.Structural insights into VAChT neurotransmitter recognition and inhibition[J].Cell Research, 2024.
• Chunyu J, Chengzhi Y, et al.A new anti-CRISPR gene promotes the spread of drug-resistance plasmids in Klebsiella pneumoniae[J].Nucleic Acids Research, 2024.
• You L, Omollo E O, Yu C, et al. Structural basis for intrinsic transcription termination[J]. Nature, 2023.
• Huang J, Yang L, Yang L, et al. Stigma receptors control intraspecies and interspecies barriers in Brassicaceae[J]. Nature, 2023.
• Jiang L, Xie X, Su N, et al. Large Stokes shift fluorescent RNAs for dual-emission fluorescence and bioluminescence imaging in live cells[J]. Nature Methods, 2023.
• Bai X , Sun P , et al. Zhe.Structure and dynamics of the EGFR/HER2 heterodimer[J].cell discovery, 2023.
使用Trans1-T1 Phage Resistant Chemically Competent Cell (CD501)产品发表的部分文章:
• Zhang R W, Zhou H, et al. Amplification editing enables efficient and precise duplication of DNA from short sequence to megabase and chromosomal scale [J]. Cell, 2024.
• Wang C, Wang J, Lu J, et al. A natural gene drive system confers reproductive isolation in rice[J]. Cell, 2023.
• Shan L, Xu G, Yao R W, et al. Nucleolar URB1 ensures 3′ ETS rRNA removal to prevent exosome surveillance[J]. Nature, 2023.
• Luo Y, Liu S, Xue J, et al. High-throughput screening of spike variants uncovers the key residues that alter the affinity and antigenicity of SARS-CoV-2[J]. Cell Discovery, 2023.
• Lei Z, Meng H, Liu L, et al. Mitochondrial base editor induces substantial nuclear off-target mutations[J]. Nature, 2022.
• Zhang Q, Zhang X, Zhu Y, et al. Recognition of cyclic dinucleotides and folates by human SLC19A1[J]. Nature, 2022.
• Li Y, Zhao L, Zhang Y, et al. Structural basis for product specificities of MLL family methyltransferases[J]. Molecular Cell, 2022.
• Wang D, Xu C, Yang W, et al. E3 ligase RNF167 and deubiquitinase STAMBPL1 modulate mTOR and cancer progression[J]. Molecular cell, 2022.





