文章信息
文章题目:A Zea genus-specific micropeptide controls kernel dehydration in maize
期刊:Cell
发表时间:2024年11 月12日
主要内容:华中农业大学严建兵团队在Cell杂志上发表了文章A Zea genus-specific micropeptide controls kernel dehydration in maize,该研究首次揭示了玉米籽粒脱水的分子机制,鉴定到一个影响籽粒脱水的小肽microRPG1,是玉米及其近缘种中特有的一种编码31个氨基酸的新型小肽,由非编码序列从头起源,通过精确调节乙烯信号通路关键基因的表达来控制籽粒脱水。研究结果为快脱水宜机收玉米培育奠定了重要基础。
原文链接:https://www.cell.com/cell/fulltext/S0092-8674(24)01212-1
使用TransGen产品:
Trans1-T1 Phage Resistant Chemically Competent Cell (CD501)
EasyScript® One-Step gDNA Removal and cDNA Synthesis SuperMix (AE311)

研究背景
玉米作为我国种植面积最大、总产量最高的作物,由于长期受限于缺乏快脱水的品种,导致玉米籽粒机械化收获面积还不到15%,影响了生产效率和种植成本。迄今为止,控制籽粒脱水速率这一性状的基因很少被克隆,其潜在机制尚不清晰,这是难以通过遗传改良培育快脱水宜机收玉米品种的根本原因。
文章概述
首先,研究团队利用籽粒脱水表型田间鉴定技术,通过QTL定位,定位到四个影响籽粒脱水的QTL。该研究发现其中一个主效QTL-qKDR1是一段不编码任何蛋白也不转录的DNA序列,在目标区域双亲之间存在一个约6.2Kb的转座子序列。敲除双亲序列后,不管是否含有转座子,都能导致籽粒脱水速率显著降低。分析表明,qKDR1可能作为一个抑制子,抑制其上游约10Kb处一个名为RPG (qKDR1 REGULATED PEPTIDE GENE)基因的表达。进一步研究发现,RPG就是qKDR1调控的目标基因,两个转录因子ZmMYBST1和ZmMYBR43可以结合到qKDR1而抑制RPG的表达。
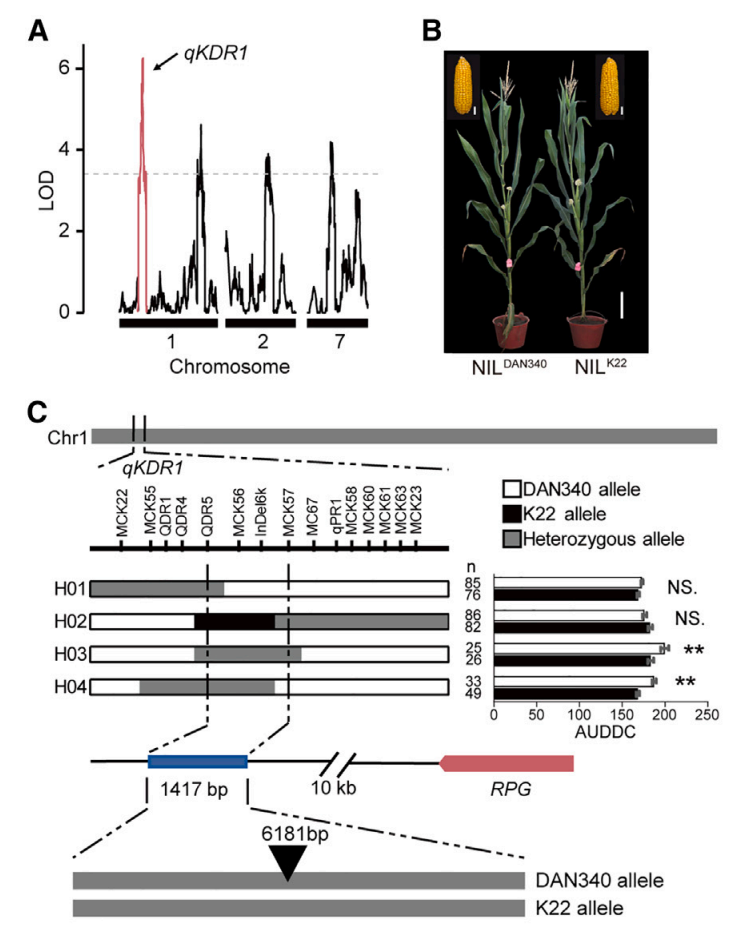
qKDR1是控制籽粒脱水的位点
RPG在玉米基因组中是一个全新的基因。研究团队通过多种技术共同证明了RPG通过编码一段31个氨基酸的小肽发挥功能,该小肽被命名为microRPG1。敲除microRPG1可加快脱水速率,超表达则显著降低脱水速率。
进一步研究发现,microRPG1可能通过调控乙烯信号途径中的关键基因ZmEIL1和ZmEIL3的表达而影响脱水。RPG在授粉后26天的籽粒中表达,在38天达到最高,此时玉米籽粒灌浆基本结束,调控乙烯的表达可以促进籽粒的快速脱水,又不影响产量,实现了产量和脱水的平衡。
microRPG1和任何已知的小肽并不同源,在其它物种中也未被鉴定到,是玉米及其近缘种特有的。研究发现,该小肽仅在玉蜀黍属和摩擦禾属中存在同源序列,在禾本科的其他成员中没有。在玉蜀黍属中,一个核苷酸(ACG到ATG)的突变产生了新的起始密码子,导致一段非编码序列起始翻译,从头产生了一个新基因。系统发育树表明,该突变可能发生在65万年前玉蜀黍属和摩擦禾属分化之后,该发现为新基因的起源提供了一个新的范例,也为从头创造新基因提供了方向。
这样一个独特的小肽在其它物种中是否起作用呢?
研究团队体外合成该小肽并外源施加于拟南芥,发现可以显著延迟角果的成熟,并显著提高了拟南芥种子的水分含量。在拟南芥中超表达microRPG1,也能够显著延迟角果的成熟。拟南芥的根可以吸收小肽并可以被运输到地上部分。这些结果暗示了microRPG1小肽在其它物种中可能具有保守的功能,这为进一步探索该小肽的应用价值提供了富有想象的空间。
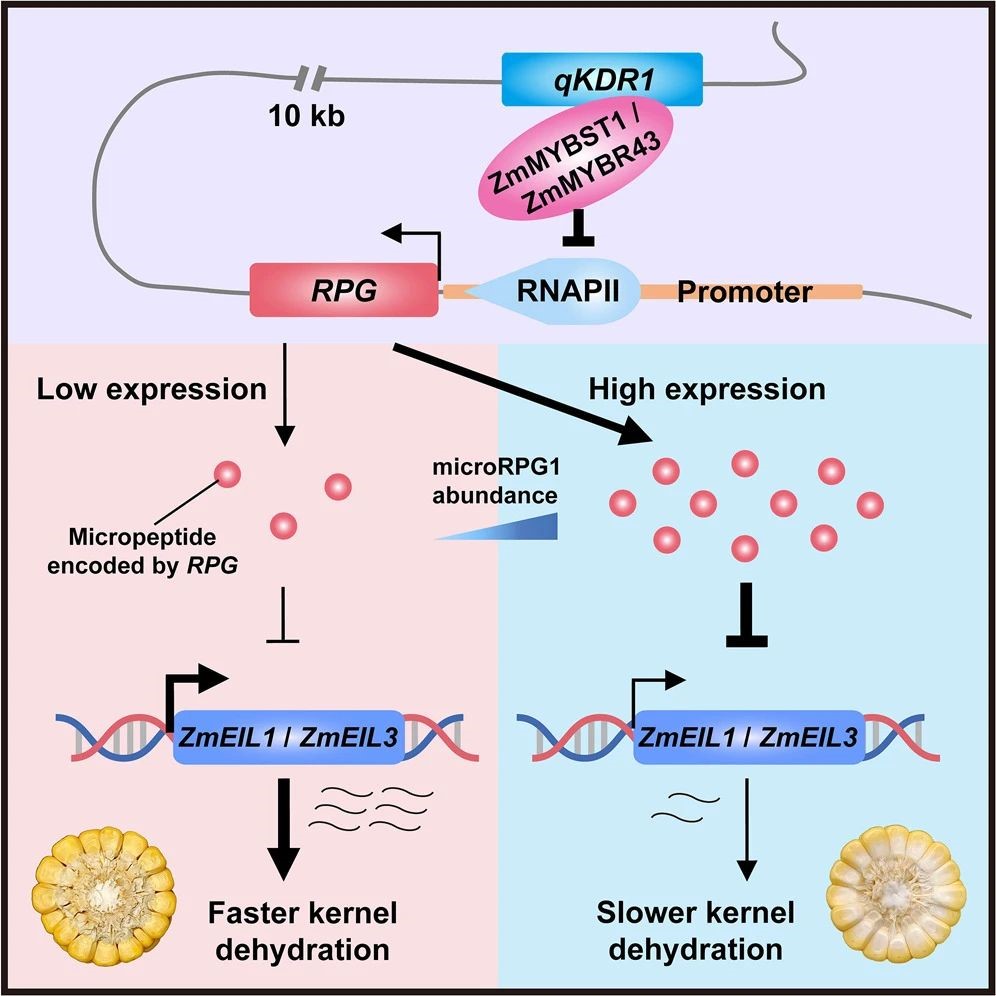
microRPG1调控籽粒脱水的机制
适合机械化收获的玉米籽粒含水量要求在15%-25%之间,但我国大多数玉米品种在收获时的含水量通常在30%-40%之间。多年多点的试验表明,敲除microRPG1可使收获时的籽粒含水量下降2%-17%,平均下降7%,同时其他农艺和产量性状没有明显的变化。研究团队分析了数百份具有代表性的玉米种质材料,发现几乎所有的材料都存在RPG基因,这意味着操纵RPG来改变籽粒脱水速率培育宜机收的品种具有巨大的应用潜力。据悉,团队围绕玉米籽粒脱水的精准调控已经布局多个专利,并授权未米生物公司开展商业化应用,目前已经取得良好进展。
全式金生物产品支撑
优质的试剂是科学研究的利器。全式金生物的克隆感受态细胞Trans1-T1 Phage Resistant Chemically Competent Cell (CD501)和RT-PCR产品EasyScript® One-Step gDNA Removal and cDNA Synthesis SuperMix (AE311)助力本研究。
Trans1-T1 Phage Resistant Chemically Competent Cell (CD501)
本产品经特殊工艺制作,可用于DNA的化学转化。使用pUC19质粒DNA检测,转化效率高达109 cfu/μg DNA以上,自上市以来多次荣登Cell和Nature等知名期刊,助力科学研究。
产品特点:
• 生长速度快,在氨苄青霉素平板上,8-9 小时可见克隆。
• 用于蓝、白斑筛选,12 小时可见蓝斑。
• 将过夜培养的单克隆在2 mL的LB培养基中培养4-5小时即可进行小量质粒提取。
• 适用于高效的DNA 克隆和质粒扩增,减少克隆DNA同源重组的发生,提高质粒DNA的产量和质量。
• 具有T1,T5噬菌体抗性。
EasyScript® One-Step gDNA Removal and cDNA Synthesis SuperMix (AE311)
本产品以RNA为模板,在同一反应体系中,合成第一链cDNA的同时去除RNA模板中残留的基因组DNA。反应结束后,只需在85℃加热5秒钟,即可同时失活EasyScript® RT/RI与gDNA Remover,自上市以来多次荣登Cell、Nature和Science等知名期刊,助力科学研究。
产品特点:
• 在同一反应体系中,同时完成反转录与基因组DNA的去除,操作简便,降低污染机率。
• 产物用于qPCR:反转录15分钟;产物用于PCR:反转录30分钟。
• 将反应结束后,同时热失活RT/RI与gDNA Remover。与传统的用DNase I预处理RNA的方法相比,避免了处理后热失活DNase I对RNA的损伤。
• 操作简单。
• 合成片段≤8 kb。
全式金生物产品再一次登上Cell期刊,证明了大家对全式金生物产品品质和实力的认可,也完美诠释了全式金生物一直以来秉承的“品质高于一切,精品服务客户”的理念。全式金生物始终在助力科研的道路上砥砺前行,希望未来能与更多的科研工作者并肩奋斗,用更多更好的产品持续助力科研。
使用Trans1-T1 Phage Resistant Chemically Competent Cell (CD501)产品发表的部分文章:
• Yu Y H, Li W Q, Liu Y F, et al. A Zea genus-specific micropeptide controls kernel dehydration in maize [J]. Cell, 2024.
• Zhang R W, Zhou H, et al. Amplification editing enables efficient and precise duplication of DNA from short sequence to megabase and chromosomal scale [J]. Cell, 2024.
• Wang C, Wang J, Lu J, et al. A natural gene drive system confers reproductive isolation in rice[J]. Cell, 2023.
• Shan L, Xu G, Yao R W, et al. Nucleolar URB1 ensures 3′ ETS rRNA removal to prevent exosome surveillance[J]. Nature, 2023.
• Luo Y, Liu S, Xue J, et al. High-throughput screening of spike variants uncovers the key residues that alter the affinity and antigenicity of SARS-CoV-2[J]. Cell Discovery, 2023.
• Lei Z, Meng H, Liu L, et al. Mitochondrial base editor induces substantial nuclear off-target mutations[J]. Nature, 2022.
• Zhang Q, Zhang X, Zhu Y, et al. Recognition of cyclic dinucleotides and folates by human SLC19A1[J]. Nature, 2022.
• Li Y, Zhao L, Zhang Y, et al. Structural basis for product specificities of MLL family methyltransferases[J]. Molecular Cell, 2022.
• Wang D, Xu C, Yang W, et al. E3 ligase RNF167 and deubiquitinase STAMBPL1 modulate mTOR and cancer progression[J]. Molecular cell, 2022.
使用EasyScript® One-Step gDNA Removal and cDNA Synthesis SuperMix (AE311)产品发表的部分文章:
• Yu Y H, Li W Q, Liu Y F, et al. A Zea genus-specific micropeptide controls kernel dehydration in maize [J]. Cell, 2024.
• Shi Q, Xia Y, Xue N, et al. Modulation of starch synthesis in Arabidopsis via phytochrome B‐mediated light signal transduction[J]. Journal of Integrative Plant Biology, 2024.
• Wu K, Wang S, Song W, et al. Enhanced sustainable green revolution yield via nitrogen-responsive chromatin modulation in rice[J]. Science, 2020.
• Li S, Tian Y, Wu K, et al. Modulating plant growth–metabolism coordination for sustainable agriculture[J]. Nature, 2018.
• Lin J L, Chen L X, Wu W K, et al. Single-cell RNA sequencing reveals a hierarchical transcriptional regulatory network of terpenoid biosynthesis in cotton secretory glandular cells[J]. Molecular plant, 2023.
• Lin J L, Fang X, Li J X, et al. Dirigent gene editing of gossypol enantiomers for toxicity-depleted cotton seeds[J]. Nature Plants, 2023.
• Sheng C, Zhao J, Di Z, et al. Spatially resolved in vivo imaging of inflammation-associated mRNA via enzymatic fluorescence amplification in a molecular beacon[J]. Nature Biomedical Engineering, 2022.
• Mo J, Chen Z, Qin S, et al. TRADES: targeted RNA demethylation by suntag system[J]. Advanced Science, 2020.





